Molekulardynamiken komplexer Proteine entschlüsseln
Hochauflösende NMR-Spektroskopie kombinert mit Computer-Simulationen liefert Einblick in räumliche Protein-Strukturen.
Proteine bestehen aus Aminosäuren, die nach Vorgabe des jeweiligen Erbguts zu langen Aminosäureketten verknüpft werden. Diese liegen jedoch nicht einfach wie Perlenketten zusammengerollt in den Zellen vor, sondern falten sich zu komplexen, dreidimensionalen Strukturen. Dabei bestimmt die Faltung eines Proteins in entscheidendem Maße dessen Funktion. Aus ihr geht beispielsweise hervor, mit welchen anderen Molekülen ein Protein in der Zelle wechselwirken kann. Die Kenntnis über die räumliche Struktur von Proteinen ist daher von großem Interesse für die Lebenswissenschaften und spielt unter anderem bei der Entwicklung von Arzneimitteln eine Rolle.
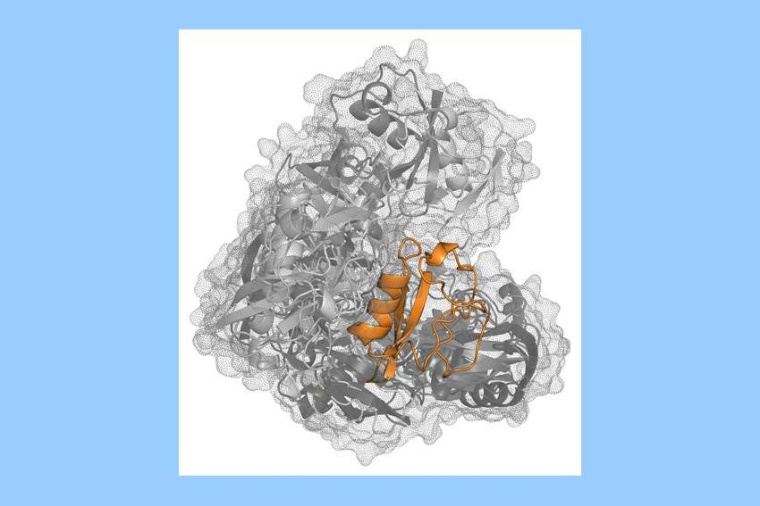
„Leider ist die Aufklärung der räumlichen Struktur eines Proteins alles andere als trivial und die Fokussierung auf einen einzelnen Zustand nicht immer zielführend, insbesondere je wandelbarer das Protein hinsichtlich seiner Struktur ist“, so Tobias Schneider von der Uni Konstanz. Der Grund: Komplexe Proteine falten sich häufig in mehrere kompakte Untereinheiten, Domänen genannt, die wiederum durch flexible Elemente miteinander verbunden sein können. Je mehr dieser flexibel verbundenen Untereinheiten es gibt, desto mehr mögliche dreidimensionale Strukturen kann ein Protein theoretisch annehmen. „So kann es passieren, dass ein Protein in Lösung, zum Beispiel im Inneren unserer Zellen, mehrere gleichberechtigte Zustände besitzt und ständig zwischen diesen wechselt“, erklärt Schneider.
Eine einfache Momentaufnahme reicht also nicht aus, um die Struktur derartiger Multidomänen-Proteine vollständig zu beschreiben, da diese jeweils nur einen von vielen Zuständen erfassen würde. Um ein detailliertes Bild der möglichen räumlichen Strukturen solcher Proteine zu erhalten, bedürfte es vielmehr der geschickten Kombination verschiedener Methoden. Einen entsprechenden Ansatz unter Verwendung sich gegenseitig ergänzender Methoden haben Schneider und weitere Biophysiker der Uni Konstanz jetzt vorgestellt.
„Die NMR-Spektroskopie erlaubt es zum Beispiel, Informationen über die dynamischen Eigenschaften solcher Proteine zu gewinnen. Aufwändige Computersimulationen geben hingegen eine gute Übersicht über die Bandbreite möglicher Faltungen“, erläutert Team-Mitglied Michael Kovermann. „Eine Lösung, welche die dynamischen und strukturellen Eigenschaften von Multidomänen-Proteinen umfänglich abbildet, fehlte bisher.“ Die Forscher erarbeiteten daher einen Arbeitsablauf, der NMR-Spektroskopie und Computersimulationen miteinander verknüpft, sodass Informationen zu beiden Eigenschaften mit hoher zeitlicher und räumlicher Auflösung gewonnen werden können.
Den Nachweis, dass die Methode funktioniert, lieferten die Forscher gleich mit. Sie untersuchten hierfür verschiedene Ubiquitin-Dimere. Diese bestehen aus zwei Einheiten des Proteins Ubiquitin, die durch eine flexible Bindung miteinander verknüpft sind, ähnlich wie es auch in der Zelle vorkommt. Es handelt sich also um ein Paradebeispiel für ein Multidomänen-Protein, für das bisher unterschiedliche Strukturmodelle vorgeschlagen wurden und das von großem wissenschaftlichen Interesse ist.
Die Forscher konnten zeigen, dass die untersuchten Ubiquitin-Dimere eine hohe strukturelle Variabilität aufweisen und dass diese mit der entwickelten Methodenkombination detailliert beschrieben werden kann. Somit erklären die Ergebnisse auch die unterschiedlichen Strukturmodelle, die für Ubiquitin-Dimere bislang existieren.
„Wir sind überzeugt, dass unser Ansatz – die Kombination sich ergänzender Methoden – nicht nur bei Ubiquitin-Dimeren, sondern auch bei anderen Multidomänen-Proteinen funktioniert“, so Schneider. „Damit eröffnet unsere Studie neue Wege zu einem besseren Verständnis der hohen Strukturvielfalt, die diesen komplexen Proteinen innewohnt und eine entscheidende Rolle für ihre biologischen Funktionen spielt.“
U. Konstanz / RK
Weitere Infos
- Originalveröffentlichung
T. Schneider et al.: Specifying conformational heterogeneity of multi-domain proteins at atomic resolution, Structure, online 8. August 2023; DOI: 10.1016/j.str.2023.07.008 - Magnetische Resonanzspektroskopie (M. Kovermann), Biophysik, Universität Konstanz